
Modelado computacional del sistema inmune
Laura Vargas-Parada
17 de abril de 2019
“Esta imagen integra dos niveles de organización, una visión molecular y una visión celular de la vida”, dijo Luis Mendoza, experto en modelado y simulación de redes reguladoras de interés biológico, al mostrar una acuarela de David S. Goodsell de una célula de Mycoplasma mycoides y todas las macromoléculas que la componen. “Yo estoy tratando de hacer lo mismo [integrar los dos niveles de organización], pero a partir de las matemáticas”.
La idea, explicó el académico adscrito al Instituto de Investigaciones Biomédicas de la UNAM, es no sólo enfocarse en una molécula específica sino en cómo muchas moléculas interactúan entre sí para dar lugar a ciertos comportamientos celulares muy particulares y cómo estas interacciones moleculares determinan de alguna forma cuál es el comportamiento de las células y cómo interactúan entre sí.
Mendoza impartió la primera sesión aplicada Immune Systems Computational Modeling: From the Construction of Genetic Regulatory Networks to Virtual Cultures dentro del curso ImmunoInformatics 2019 el pasado 8 de abril en el Centro de Ciencias de la Complejidad (C3) de la UNAM.
Biología de sistemas

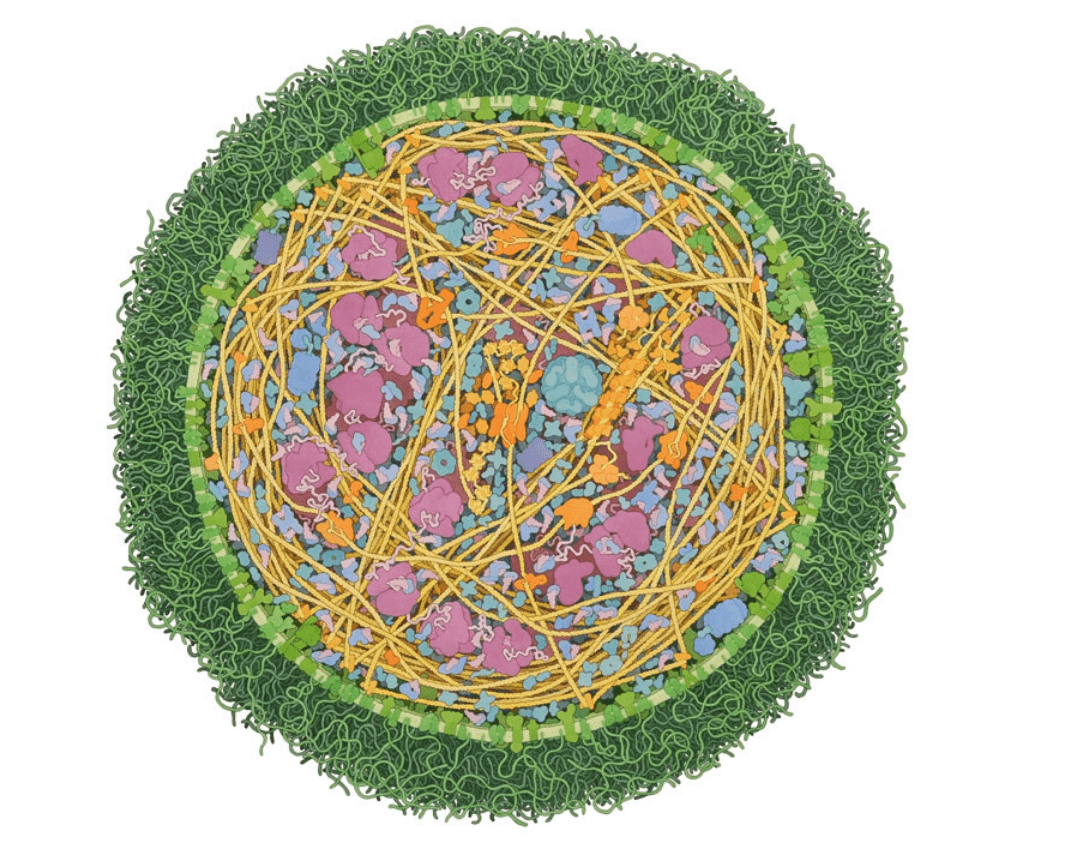
Acuarela de David S. Goodsell de una célula de Mycoplasma mycoides
http://mgl.scripps.edu/people/goodsell/illustration/mycoplasma
Al comenzar su conferencia el académico explicó que su tema no estaría enfocado a la inmunoinformática sino a la biología de sistemas ya que la mayor parte de su investigación tiene que ver con la integración de niveles de organización. “Estamos tratando de comprender cuáles son las propiedades generales de algunas redes”, dijo a un auditorio completamente lleno.
La biología de sistemas explicó, busca estudiar simultáneamente diferentes niveles de información biológica, células que interactúan entre sí y forman tejidos, los tejidos que en su conjunto forman órganos y los organismos que interactúan entre sí. De forma que hay muchos niveles de organización en los cuales uno puede tratar de comprender el comportamiento de un sistema. Por tanto, se puede entender a la biología de sistemas como una biología de redes.
Para Lenin Domínguez Ramírez, doctor en Ciencias Bioquímicas y especialista en bioinformática estructural de la Universidad de las Américas en Puebla la biología de sistemas es la versión real de un sueño que comparten los físicos posteriores a Newton, “poder usar ecuaciones simples parar no solo medir sino predecir el comportamiento de sistemas complejos. La biología de sistemas permite sintetizar el conocimiento de áreas como la bioquímica, biología molecular y celular generando modelos representativos a diferentes niveles de complejidad”, escribió en un mensaje de correo electrónico.
Mendoza, doctor en Ciencias Biomédicas, explicó durante su ponencia que, en su caso, el sistema que le interesa estudiar es la hematopoyesis, “proceso que genera todas las líneas celulares de la sangre y que comienza en la médula ósea con diferentes interacciones moleculares y celulares. Lo que hoy voy a presentarles está en esos dos niveles de organización, molecular y celular, con el objetivo de mostrarles que es posible ver el comportamiento de algunos subsistemas del sistema inmune a partir de metodologías teóricas”.
Redes
Cuando se integran los datos moleculares provenientes de la investigación experimental es posible construir esquemas que muestren cascadas o redes de interacción entre genes que son muy útiles para conocer qué molécula activan o inhiben a otra.
Sin embargo, estas redes tienen la limitante de que son estáticas, esto es, están los principales elementos del proceso “pero no conocemos cuáles son las restricciones o qué tipo especial de patrones se pueden obtener con estas interacciones. Esto no puede obtenerse sólo viendo la conectividad, falta una dimensión aquí, una dimensión temporal”, explicó el académico y agregó: “lo que les voy a mostrar son algunas herramientas específicas para analizar el comportamiento dinámico de redes de interacción entre genes”.
En este tipo de redes el enfoque se centra en el comportamiento dinámico de los elementos que componen la red. La idea es tratar de comprender el comportamiento global del sistema yendo de lo específico, los genes, a lo general, el comportamiento del sistema.
Es aquí donde entra la complejidad, que se basa en comprender que el todo es más que la suma de sus partes. El Institute for Systems Biology de Seattle, EUA, describe la Biología de Sistemas como una disciplina que “adopta la complejidad biológica y se embarca a descifrar grandes cantidades de datos para obtener información valiosa y lograr avances entre disciplinas científicas”.
De los modelos a los pronósticos: modelado computacional de la hematopoyesis
Mendoza revisó durante su presentación dos metodologías generales para analizar las interacciones moleculares. La primera fue la construcción de redes booleanas, donde solo hay dos posibles estados para cada nodo o unidad funcional de la red (0 o 1, activo o inactivo, ausente o presente) y cómo asignar reglas matemáticas que permitan representar cómo se regulan la interacción entre moléculas. En este tipo de modelo el tiempo es una unidad discreta, no es un continuo como en la vida real, sino una simplificación en la que puede verse por etapas discretas, tiempo 1, tiempo 2, tiempo 3, etc.
En su presentación mostró cómo pueden integrarse varios modelos para generar una red de interacciones y cómo con estas pequeñas redes regulatorias es posible “representar uno de los principales elementos de la diferenciación celular que involucra patrones estables de activación que van de una rama a otra del proceso general”.
En su trabajo de investigación, Mendoza ha analizado diferentes aspectos del proceso de diferenciación en la hematopoyesis como el comportamiento dinámico de distintas subredes, por ejemplo, la red que controla la regulación de la diferenciación de células NK o natural killers. “La belleza de estos modelos es que […] es posible pronosticar la existencia de algunas interacciones o intermediarios” que aún no han sido descritos experimentalmente, explicó. “Esta es la importancia de incorporar aspectos dinámicos en el modelado”.
Para Jennifer Enciso, embajadora mexicana de Immunopaedia, una de las instancias organizadoras del evento junto con la International Union of Immunological Societies (IUIS), la Asociación Latinoamericana de Inmunología (ALAI), la Sociedad Mexicana de Inmunología (SMI) y el propio C3, consideró que el trabajo que Mendoza ha desarrollado es muy relevante por la integración que ha logrado de las evidencias experimentales respecto a la diferenciación de células hematopoyéticas. “Estos modelos implican una ardua tarea de curación manual y un extensivo análisis de propiedades dinámicas”, dijo en entrevista por mensajería instantánea.
El ejemplo que Mendoza presentó sobre células NK es muy reciente y aún no ha habido confirmación de los pronósticos derivados del modelo, pero el investigador mostró otro estudio donde los pronósticos teóricos fueron luego confirmados de forma experimental. Mostró además otro trabajo experimental que está en vías de publicación y en el que se utilizan el mismo tipo de redes para estudiar las redes de regulación entre células dendríticas.
Sin embargo, existen otros procesos que presentan un reto para el modelo booleano, por ejemplo, estudiar la activación del receptor de células T durante el proceso de diferenciación. Para este caso, el académico presentó una segunda metodología de trabajo basada en modelos continuos que permiten pasar de interacciones con solo dos valores posibles (0 o 1) a un gradiente que incluyan un número infinito de estados posibles.
Finalmente, el investigador introdujo la idea de modelar la interacción entre distintas células. “Cada célula puede estudiarse individualmente y luego ver el cultivo celular como una red de redes. Básicamente, lo que uno hace es un modelo de compartimentos”, concluyó.